比较基因组方法揭示深海灰蝾螺适应深海环境及其进化的分子机制研究获新进展
中国科学院海洋研究所发布了深海灰蝾螺高质量染色体水平基因组参考序列,基于比较基因组方法探索驱动深海灰蝾螺适应深海热液喷口的分子机制,成果2024年11月10日在学术期刊Science of The Total Environment正式发表。
深海灰蝾螺Thermocollonia jamsteci(Mollusca、Gastropoda、Vetigastropod、Trochida、Colloniidae)是深海热液喷口中具有代表性的腹足动物。深海热液喷口的特点是高静压、缺氧、黑暗和有毒物质,目前人们对生物体如何适应这种极端的海洋生态系统仍然知之甚少。本研究利用二代DNA测序和三代DNA测序技术完成了对深海灰蝾螺基因组的预测和组装,利用Hi-C技术完成了灰蝾螺染色体组装,构建了深海灰蝾螺染色体水平基因组参考序列。组装得到的基因组大小为2.54Gb,Contig N50为1.16Mb,Scaffold N50为231.02 Mb,组装结果成功挂载到11条染色体上,挂载率为96.68%。比较分析发现深海灰蝾螺染色体经历了大规模重排、反转、融合与混合。
研究团队利用从头注释技术、基于同源基因的注释技术,并利用二代转录组和全长转录组测序数据,完成了灰蝾螺基因的蛋白编码基因注释。利用这些基因组及其注释结果,通过比较基因组技术研究了深海灰蝾螺对深海适应性进化的分子机制,为与深海适应相关的遗传变化提供了新的见解。深海灰蝾螺基因组大小是迄今为止Vetigastropoda亚纲动物基因组中最大的,影响基因组大小变异的因素包括基因组复制事件、单个基因或基因家族的丢失或获得、重组事件、转座子扩张。
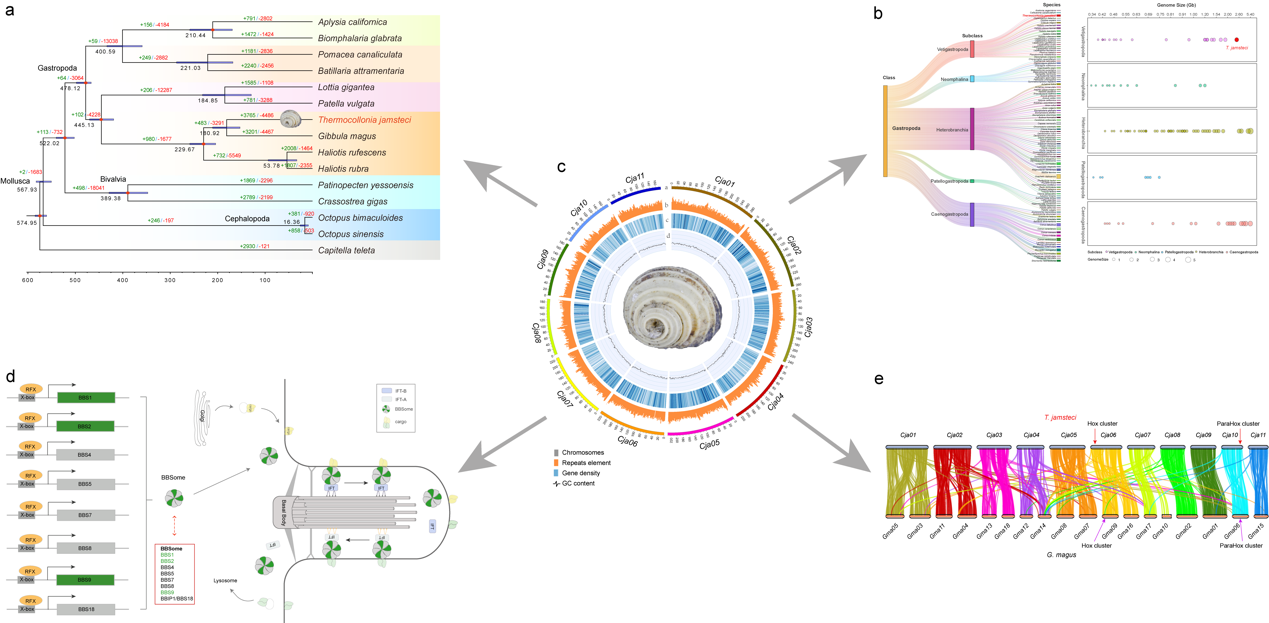
图 深海灰蝾螺比较基因组分析
a)15个软体动物的系统发育树;b)不同腹足类谱系中具有基因组的物种分布;c)深海灰蝾螺基因组特征;d)深海灰蝾螺中正选择的纤毛基因(BBS1、BBS2和BBS9)和转录因子基因RFX4;e)深海灰蝾螺和其近缘物种Gibbula maguss染色体的比较分析
染色体内基因顺序在长时间跨度内的混乱是无数次重叠倒位和其他染色体内重排的累积效应。系统发育分析表明,深海灰蝾螺分化于180.92Ma(95%的置信区间为153.88-210.48 Ma),这和当时发生的三叠纪末(~200Ma)大规模灭绝有很大关系。BBSome和RFX4 基因在深海灰蝾螺中呈正选择,它们除了在纤毛蛋白运输方面发挥着关键作用,也在纤毛介导的信号转导中起作用,调控着发育和机体的稳态。在深海高压环境下,深海灰蝾螺通过增强纤毛内运输机制来抵抗深海高压和缺氧环境。总之,对深海灰蝾螺的基因组大小和染色体进化的分析,以及所涉及的纤毛内运输机制,结合地质史上的三叠纪末期大规模灭绝事件,为研究深海软体动物的适应性进化机制提供了新的维度。
中国科学院海洋生态与环境科学重点实验室博士后段泽林为论文第一作者,通讯作者包括陈楠生研究员和惠敏副研究员。王静、刘淑雅、徐青、陈浩和李超伦为该论文共同作者。本项目得到了中国科学院战略性先导科技专项、国家自然科学基金和崂山实验室等项目资助。
论文信息:
Zelin Duan, Jing Wang, Shuya Liu, Qing Xu, Hao Chen, Chaolun Li, Min Hui*, Nansheng Chen*. Positive selection in cilia-related genes may facilitate deep-sea adaptation of Thermocollonia jamsteci[J]. Science of The Total Environment, 2024, 950: 175358.
https://doi.org/10.1016/j.scitotenv.2024.175358.
附件下载:
 鲁公网安备37020202001323号
鲁公网安备37020202001323号 | 古镇口园区地址:青岛市西海岸新区海军路88号 南海路园区地址:青岛市市南区南海路7号 科考船码头基地:青岛市西海岸新区长江东路8号 |
邮编:266000 邮件:iocas@qdio.ac.cn 电话:0532-82898611 传真:0532-82898612 |
