单株系宏条形码分析揭示骨条藻物种基因组内核糖体基因序列多样性
中国科学院海洋研究所陈楠生课题组通过对骨条藻物种单株系开展基于核糖体基因18S rDNA V4区的宏条形码分析,发现每个骨条藻单株系内存在大量18S rDNA V4扩增子序列变异(amplicon sequence variants,ASVs),表明其基因组内存在较高水平的rDNA基因组内变异(intra-genomic variation,IGV)。这项成果近期发表在学术期刊Environmental Research(Top期刊Q1,IF=7.7)。
骨条藻属(Skeletonema)物种是我国近海的优势浮游植物,也是海洋生态系统中的重要初级生产者;多种骨条藻物种可引发赤潮,危害海洋生态环境。生态调查发现骨条藻属物种在我国广泛分布。基于通用分子标记18S rDNA V4的宏条形码分析发现骨条藻具有较高的分子多样性(molecular diversity),且远远高于物种多样性(species diversity)。浮游植物的rDNA由一段段基因簇串联重复构成的,每个基因簇(拷贝单元)由基因间隔区和18S、ITS1、5.8S、ITS2和28S rDNA构成,拷贝单元之间由于协同进化高度相似,然而高度相似性并不意味着完全的序列同一性,拷贝单元之间存在变异,在真核生物中被广泛报道。因此,环境调查宏条形码方法揭示的分子多样性不仅包括了物种间的多样性和物种内的遗传多样性,还包括基因组内拷贝多样性,并且基因组内拷贝多样性占据主导贡献。由此研究团队推测骨条藻物种的18S rDNA V4也存在较高的IGVs。
研究团队选择了49个骨条藻株系(7个骨条藻物种)开展了单株系宏条形码分析,探讨宏条形码分析中高水平分子多样性的特点及其本质。结果发现每个骨条藻株系含有大量骨条藻ASVs,包括1个或者两个相对丰度很高的优势ASVs和大量的非优势ASVs,表明宏条形码分析获得的分子多样性以基因组内拷贝序列多样性(即IGVs)为主导。大多数物种的所有株系共享同一个优势ASV,因此该优势ASVs决定了物种多样性。但是,中肋骨条藻(Skeletonema costatum)的7个株系中具有两个优势ASVs,并且在不同株系中两者占比不同,表明骨条藻的优势ASVs不仅代表骨条藻物种多样性,也可能代表物种内遗传多样性。
该研究以基因组内核糖体基因18S rDNA V4序列变异为切入点,开展单株系宏条形码分析,探讨宏条形码分析中高水平分子多样性的特点及其本质,为环境样本的宏条形码分析调查提供正确的解读思路。
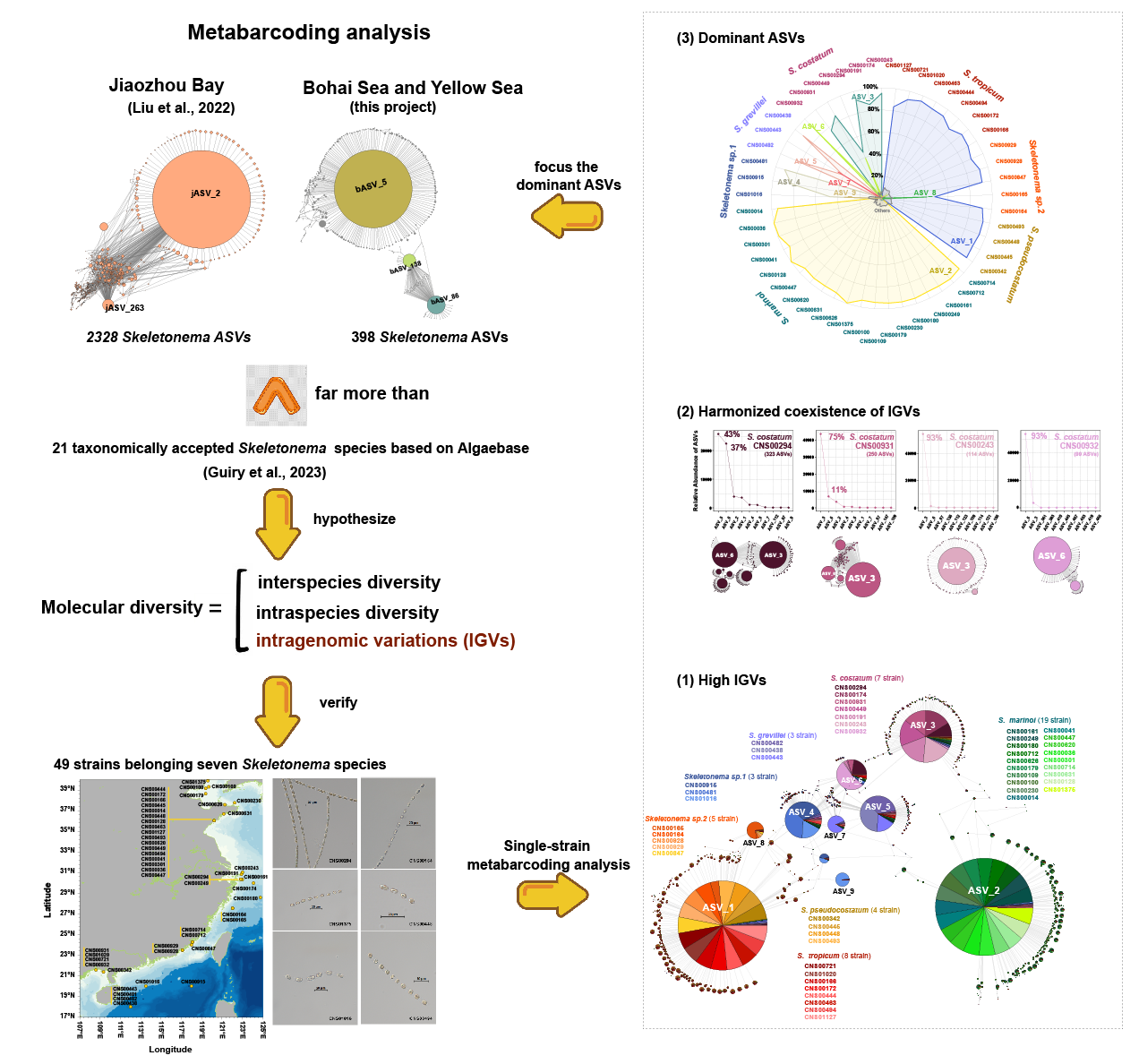
骨条藻属单株系宏条形码分析揭示基因组内变异
该论文第一作者是中国科学院海洋生态与环境科学重点实验室特别研究助理刘淑雅博士,通讯作者是研究员陈楠生。博士研究生丁翔翔和刘奎艳也参与了这部分工作。相关研究得到国家重点研发计划、自然科学基金委面上基金和青年基金项目等项目资助。
相关文章及链接:
Shuya Liu,Xiangxiang Ding,Kuiyan Liu,Nansheng Chen*,Harmonized coexistence of intragenomic variations in diatom Skeletonema strains,Environmental Research,Volume 262,Part 1,2024,119799,https://doi.org/10.1016/j.envres.2024.119799.
附件下载:
 鲁公网安备37020202001323号
鲁公网安备37020202001323号 | 古镇口园区地址:青岛市西海岸新区海军路88号 南海路园区地址:青岛市市南区南海路7号 科考船码头基地:青岛市西海岸新区长江东路8号 |
邮编:266000 邮件:iocas@qdio.ac.cn 电话:0532-82898611 传真:0532-82898612 |
