海洋所在近海抗生素抗性基因研究中取得新进展
近日,中国科学院海洋研究所海藻遗传与发育团队在环境科学期刊Environmental Pollution发表题为“Metagenomic analysis reveals the composition and sources of antibiotic resistance genes in coastal water ecosystems of the Yellow Sea and Yangtze River Delta”的研究论文。该研究基于宏基因组学,揭示了黄海及长江三角洲海域中抗生素抗性基因(ARGs)的多样性、空间分布特征及主要来源,为沿海生态系统的耐药性污染防控提供了重要科学依据。
抗生素的过度使用导致了细菌耐药性的发展和环境中ARGs的出现。目前,ARGs的扩散已成为全球公共卫生与生态安全的重大威胁。作为陆海交互作用的关键区域,沿海水域是陆源污染物的主要汇集地,也是ARGs通过食物链传递的潜在枢纽,但准确识别水体ARGs组成特征和环境驱动机制仍面临挑战。传统方法多依赖于qPCR或高通量qPCR,但由于受到特异性引物和已知基因的限制,难以全面解析ARGs的具体组成及其复杂来源。本研究基于宏基因组,对黄海和长江三角洲海域的ARGs、移动遗传元件(MGEs)和细菌群落进行了研究,为耐药基因的防治提供了新视角。
研究发现,多药耐药基因是最丰富的ARGs,与ARGs的水平基因转移(HGT)密切相关的转座子和insertion_element_IS91是主要的MGEs。温度、溶解氧、pH和深度影响ARGs在海水中的分布,而石油、农业、畜牧业和污水处理厂可能是ARGs的主要来源。从控制ARGs的角度来看,细菌群落对ARGs的发展贡献最大,并可能携带ARGs,沿洋流从长江三角洲海域扩散到黄海。与Tara Oceans全球采样数据集的比较显示,研究区域的主要ARG类型和属水平细菌与全球特征一致,但ARG亚型存在差异。本研究拓展了对近海ARGs分布格局的认识,为沿海生态系统的耐药性风险管理提供了科学依据。
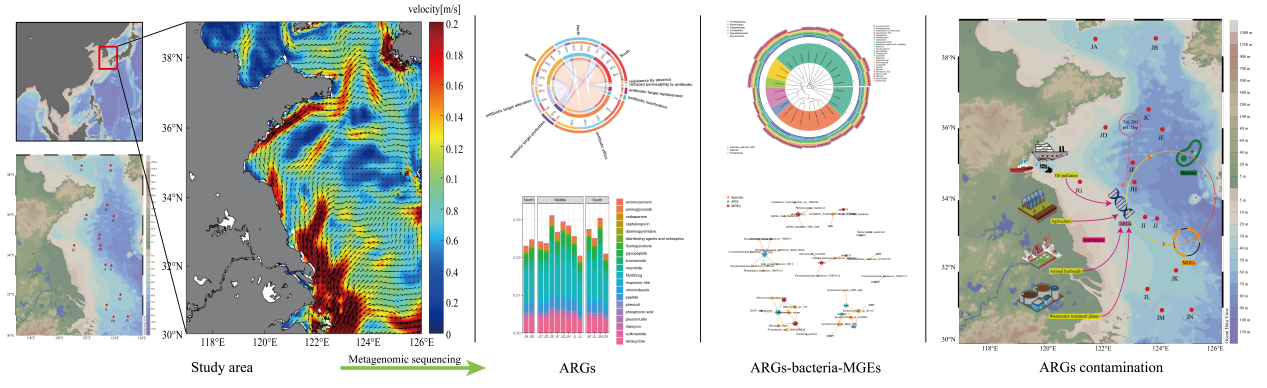
黄海和长江三角洲海域ARGs的组成、传播机制和潜在污染源
中国科学院海洋研究所硕士研究生王鑫为文章第一作者,副研究员邵展茹为通讯作者。该研究得到国家自然科学基金、中国科学院战略性先导科技专项、中国科学院青年创新促进会及农业农村部海藻类肥料重点实验室等项目的共同资助。
论文信息:
Wang,X.,Lin,Y.,Li,S.,Wang,J.,Li,X.,Zhang,D.,Duan,D.,Shao,Z.,2025. Metagenomic analysis reveals the composition and sources of antibiotic resistance genes in coastal water ecosystems of the Yellow Sea and Yangtze River Delta. Environmental Pollution,371:125923.https://doi.org/10.1016/j.envpol.2025.125923
附件下载:
 鲁公网安备37020202001323号
鲁公网安备37020202001323号 | 古镇口园区地址:青岛市西海岸新区海军路88号 南海路园区地址:青岛市市南区南海路7号 科考船码头基地:青岛市西海岸新区长江东路8号 |
邮编:266000 邮件:iocas@qdio.ac.cn 电话:0532-82898611 传真:0532-82898612 |
